
Artigo original
Sobrevivência de Salmonella enterica às condições gástricas simuladas in vitro: papel dos fatores de adesão e resistência antibacteriana
Survival of Salmonella enterica under in vitro simulated gastric conditions: role of adhesion factors and antibacterial resistance
Jhennifer Arruda Schmiedt* Emanoelli Aparecida Rodrigues dos Santos†
Emanoelli Aparecida Rodrigues dos Santos† Leonardo Ereno Tadielo‡
Leonardo Ereno Tadielo‡ Tatiane Barbosa Coitinho§
Tatiane Barbosa Coitinho§ Taís Luana Pinto¶
Taís Luana Pinto¶ Carolina Dias Rodrigues**
Carolina Dias Rodrigues** Juliano Gonçalves Pereira††
Juliano Gonçalves Pereira†† Vanessa Mendonça Soares‡‡
Vanessa Mendonça Soares‡‡ Thiago Henrique Bell駧
Thiago Henrique Bell駧 Luiz Gustavo Bach¶¶
Luiz Gustavo Bach¶¶ Vinicius Cunha Barcellos***
Vinicius Cunha Barcellos*** Luciano dos Santos Bersot†††
Luciano dos Santos Bersot†††

https://doi.org/10.18593/evid.32846
Recebido em 17 de junho de 2023 | Aceito em 09 de outubro de 2023
Resumo:
O estudo tem como objetivo verificar a capacidade de adesão e formação de biofilme de isolados de Salmonella spp. produtores de enzimas beta-lactamases de espectro estendido (ESBL), bem como a resistência de isolados produtores ou não de ESBL, frente às condições gástricas e intestinais simuladas in vitro. Foram utilizados 18 isolados de Salmonella spp. obtidos de cortes de frangos congelados produzidos no Estado do Paraná, Brasil. Os isolados produtores de ESBL foram avaliados de forma fenotípica e genotípica quanto a capacidade de adesão e formação de biofilme em lâminas de polipropileno. Além disso, todos os isolados foram submetidos a avaliação in vitro de resistência às condições gástricas e intestinais e após as simulações, realizadas contagens das células sobreviventes. Com relação à adesão e formação de biofilme, 13 isolados apresentaram essa capacidade. Quanto a tolerância às condições gástricas e intestinais, a quantificação de Salmonella spp. diminuiu (p<0,05) conforme a passagem do trato gastrointestinal. Em relação a comparação de isolados produtores e não produtores de ESBL, não houve diferença (p>0,05) na taxa de sobrevivência total. Os resultados demonstram que, apesar da diminuição da contagem de microrganismos conforme a passagem simulada pelos sucos gástricos e intestinais, a taxa de sobrevivência média total dos isolados de Salmonella spp. foi alta, o que permite inferir que os isolados possuem a capacidade de sobreviver às condições gastrointestinais simuladas, e ainda sim permaneceram em número capaz de causar infecção.
Palavras-chave: Microrganismos resistentes. Beta-lactamases. Biofilme. Tolerância gastrointestinal. Cortes cárneos. Frango de corte.
@Autor correspondente: Doutor em Ciência dos Alimentos pela Universidade de São Paulo (USP). Docente na Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil; https://orcid.org/0000-0001-7013-5574; lucianobersot@ufpr.br.
Abstract:
The study aims to assess the adhesion and biofilm formation capacity of Salmonella spp. isolates producing extended-spectrum beta-lactamases (ESBL) and the resistance of ESBL-producing and non-producing isolates to in vitro simulated gastric and intestinal conditions. Eighteen Salmonella spp. isolates were obtained from frozen chicken cuts produced in the State of Paraná, Brazil. ESBL-producing isolates were evaluated phenotypically and genotypically for their adhesion and biofilm formation capacity on polypropylene coupons. Furthermore, all isolates underwent in vitro assessment of resistance to gastric and intestinal conditions, and cell counts were conducted after the simulations. Regarding adhesion and biofilm formation, 13 isolates demonstrated this capability. Concerning tolerance to gastric and intestinal conditions, the quantification of Salmonella spp. decreased (p<0.05) as they passed through the gastrointestinal tract. When comparing ESBL-producing and non-producing isolates, there was no difference (p>0.05) in the overall survival rate. The results demonstrate that, despite the decrease in microorganism counts during simulated passage through gastric and intestinal juices, the mean overall survival rate of Salmonella spp. isolates was high. This suggests that the isolates have the capacity to survive simulated gastrointestinal conditions and remain at levels capable of causing infection.
Keywords: Resistant microrganisms. Beta-lactamases. Biofilm. Gastrointestinal tolerance. Meat cuts. Broiler chickens.
1 INTRODUÇÃO
Salmonella enterica está entre os principais patógenos envolvidos em doenças veiculadas por alimentos, sendo um grande problema econômico e de saúde pública. Estima-se a ocorrência de cerca de 93,8 milhões de casos e 155.000 mortes por ano, sendo umas das doenças mais relatadas em surtos no Brasil, União Europeia e Estados unidos1-4. A infecção é caracterizada principalmente por uma gastroenterite autolimitante qual não necessita de intervenção medicamentosa, com início dos sintomas de 6 a 72 horas após o consumo de alimento contaminado envolvendo sintomas como náuseas e vômitos, cólicas, dores abdominais e diarreias5-6. O uso de antibióticos para o tratamento só é necessário em casos mais graves da doença, como uma infecção sistêmica, que compromete principalmente pacientes idosos, crianças e imunocomprometidos7-8.
Salmonella spp. possui a habilidade de sobreviver a uma ampla faixa de temperatura e pH, além de possuir uma diversidade de sorovares, capazes de infectar humanos, animais e alimentos, sendo o frango de corte um dos principais reservatórios do microrganismo e os alimentos mais associados aos surtos são ovos e a carne de frango9-11. Os principais sorotipos associados a enfermidades transmitidas por alimentos são S. Enteritidis, S. Typhimurium e S. Infantis12-13, e em cortes cárneos de aves atualmente tem sido observado como sorotipos mais frequentes S. Typhimurium, S. Heidelberg e S. Nodolo14.
Aliado à seleção genética, esse microrganismo apresenta características de resistência aos antibióticos, produção de enzimas beta-lactamases de espectro estendido (ESBL), formação de biofilmes e resistência à sanitizantes15-16. A capacidade de produção de ESBL pode exercer vantagem na sobrevivência contra alguns antibióticos como penicilinas e cefalosporinas, contribuindo para a sobrevivência da Salmonella spp., sendo então, de extrema importância para a saúde pública. Além disso, os genes que codificam para ESBL podem estar presentes em elementos genéticos móveis, como plasmídeos, facilitando a transferência de genes entre as bactérias no ambiente intestinal, contribuindo para a propagação da resistência17.
Alguns fatores estão associados à ocorrência de salmoneloses, como o cozimento insuficiente dos alimentos, refrigeração inadequada, formação de biofilmes, tolerância dos isolados a condições de estresse, exposição aos ácidos e resistência a antibióticos e biocidas18. O estômago tem um papel fundamental na eliminação ou inativação de agentes patogênicos presentes em alimentos ou água antes de entrarem no trato intestinal19-20. O baixo pH do ambiente intragástrico pela presença de ácido clorídrico (HCl), constitui um dos principais obstáculos de defesa não específicas do corpo e a redução da secreção de ácido gástrico predispõe o organismo à infecções por uma grande variedade de microrganismos21-22. Sendo assim, para causar a doença clínica, os microrganismos devem sobreviver ao ambiente gástrico hostil, com baixo pH, e evitar a lise por sais biliares no intestino delgado superior, se anexando e invadindo o íleo distal e o cólon proximal, causando doença23.
Assim, o objetivo deste estudo foi verificar a capacidade de adesão e formação de biofilme de isolados produtores de enzimas beta-lactamases de espectro estendido, assim como a resistência de isolados de Salmonella spp. produtoras ou não de enzimas ESBL, frente às condições gástricas e intestinais simuladas in vitro, a fim de se observar a taxa de sobrevivências desses microrganismos em tais condições.
2 MATERIAL E MÉTODOS
2.1 ORIGEM DOS ISOLADOS DE SALMONELLA
Foram utilizados 18 isolados de Salmonella spp. caracterizados em estudo anterior14 a partir de cortes de frangos congelados. O isolamento foi realizado através da metodologia ISO 6579:2007 (presença ou ausência do microrganismo) e ISO 6579-2:2012 (quantificação), e a identificação dos sorotipos bacterianos foi realizada através de sorotipagem. Treze dos isolados selecionados (numeradas de 1 a 13) foram caracterizados como produtores de ESBL (Tabela 1) e cinco isolados (14 a 18) foram considerados não produtores de ESBL.
Tabela 1 – Isolados de Salmonella spp. utilizados no estudo e região do corte de frango de onde foram obtidos
|
Amostra |
Sorotipo |
Produção de ESBL |
|
1 |
Sim |
|
|
2 |
S. Heidelberg |
Sim |
|
3 |
S. Ndolo |
Sim |
|
4 |
S. Heidelberg |
Sim |
|
5 |
S. Heidelberg |
Sim |
|
6 |
S. Ndolo |
Sim |
|
7 |
S. Typhimurium |
Sim |
|
8 |
S. Typhimurium |
Sim |
|
9 |
S. Heidelberg |
Sim |
|
10 |
S. Typhimurium |
Sim |
|
11 |
S. Typhimurium |
Sim |
|
12 |
S. Ndolo |
Sim |
|
13 |
S. Typhimurium |
Sim |
|
14 |
S. Heidelberg |
Não |
|
15 |
S. Heidelberg |
Não |
|
16 |
S. Heidelberg |
Não |
|
17 |
S. Typhimurium |
Não |
|
18 |
S.Typhimurium |
Não |
Legenda: ESBL = enzimas beta-lactamases de espectro estendido
2.2 CAPACIDADE DE ADESÃO E FORMAÇÃO DE BIOFILME EM LÂMINAS DE POLIPROPILENO
A capacidade de adesão e formação de biofilmes em lâminas de polipropileno foi realizada com 13 isolados de Salmonella spp. que apresentaram a produção de ESBL. Previamente aos ensaios, as lâminas (1 cm x 1 cm x 0,1 cm) foram imersas em solução de detergente, enxaguadas com água destilada e esterilizadas em autoclave a 121°C por 15 min em frascos contendo 50 mL de caldo Luria-Bertani (LB, Oxoid®), segundo a metodologia de Tadielo et al.24. O caldo LB contendo as lâminas estéreis foi ajustado na escala 0,5 de MacFarland dos isolados testados e incubados a 37 ± 1ºC durante 96h sob agitação à 100 rpm em um agitador orbital (BIOSAN®). Como controle negativo utilizou-se um frasco não inoculado contendo lâminas estéreis e para controle positivo utilizou-se S. Typhimurium ATTC 14028, ambos incubados nas mesmas condições anteriores.
Após o período de incubação, a remoção de células viáveis das lâminas foi baseada na metodologia de Poimenidou et al.25 com adaptações. As lâminas foram retiradas da solução e lavadas com 10 mL de Tampão Fosfato-salino (PBS,do inglês Phosphate-Buffered Saline) para remoção de células planctônicas, ou seja, não aderidas. As células sésseis foram destacadas por swabs estéreis aproximadamente 100 vezes sobre a área das lâminas. Os swabs foram imersos em tubos contendo 10 mL de solução salina e homogeneizados em vortex durante 60 segundos. Após diluições decimais apropriadas, as culturas (1000 μL) foram plaqueadas em Tryptic Soy Agar (TSA, Oxoid®) e enumeradas após ٢٤h a ٣٧ºC. Os resultados foram expressos em log10 por UFC/cm2.
2.3 AVALIAÇÃO GENOTÍPICA DE ADESÃO E FORMAÇÃO DE BIOFILME
Foi realizado a extração do DNA como descrito por Dias et al.26 dos 13 isolados de Salmonella spp. Após extração, o DNA foi submetido à Reação em Cadeia da Polimerase (PCR – do inglês Polymerase Chain Reaction) para avaliação de genes relacionados a produção de fímbrias, adesão, virulência, formação de celulose e biofilme (agfD, adrA, lpfA, agfA, sipA) e quorum sensing (sidA). As reações compreenderam um volume final de 25 µL envolvendo 12,5 µL de GoTaq Green Master Mix (Promega®), ٤,٠ µL do DNA extraído, 2 μL dos primers forward e reverse (1 pmol) e 4,5 μL de Nuclease-Free Water (Promega®). As sequências dos primers, os tamanhos de fragmentos esperados e a temperatura de anelamento de cada primers estão representados na tabela 2. Os produtos de PCR foram submetidos a eletroforese em gel de agarose a 1,5% (p/v), corados com GelRed (Biotium Inc., Hayward, CA, EUA) e visualizados em transiluminador UV (Loccus Biotecnologia, SP, Brasil).
Tabela 2 – Genes alvo e sequências dos primers utilizados para caracterização molecular dos isolados de Salmonella spp.
|
Gene |
Sequência (5’->3’) |
Tamanho do produto |
Temperatura de anelamento |
Referência |
|
agfD |
F: TGCGGACTCGGTGCTGTTGT |
123 |
60°C |
Oliveira et al.27 |
|
R: CAGGAACACGTGGTCAGCGG |
||||
|
adrA |
F: GGGCGGCGAAAGCCCTTGAT |
92 |
60°C |
Oliveira et al.27 |
|
R: GCCCATCAGCGCGATCCACA |
||||
|
lpfA |
F: CTTTCGCTGCTGAATCTGGT |
250 |
55°C |
Bäumler and Heffron28 |
|
R: CAGTGTTAACAGAAACCAGT |
||||
|
agfA |
F: TCCACAATGGGGCGGCGGCG |
350 |
58°C |
Collinson et al.29 |
|
R: CCTGACGCACCATTACGCTG |
||||
|
sdiA |
F: AATATCGCTTCGTACCAC |
274 |
52°C |
Halatsi et al.30 |
|
R: GTAGGTAAACGAGGAGCAG |
||||
|
sipA |
F: CAGGGGTCGTTAGTGTATTGCGTGAGATG |
550 |
66,5°C |
Skyberg et al.31 |
|
R:CGCGTAACAAAGAACCCGTAGTGATGGATT |
2.4 MICROSCOPIA ELETRÔNICA DE VARREDURA
A microscopia eletrônica de varredura foi realizada no Laboratório de Microscopia Eletrônica de Varredura (MEV) da Universidade Federal do Paraná, Setor Palotina. As imagens foram geradas sobre lâminas de polipropileno (0,5 cm²) de cepas de S. Typhimurium que apresentaram capacidade de formação de biofilme. Os biofilmes formados sobre as lâminas de polipropileno foram fixados em solução de glutaraldeído 3% diluído em Tampão Fosfato-salino (PBS, Difco™; pH 7,2) a 4°C por 24h. Posteriormente, foram lavados três vezes com água destilada estéril por 10 minutos, após a qual iniciou-se a série de desidratação em crescentes concentrações de álcool (30, 50, 70, 80, 90, 96 e 100%) a 25°C por 10 min cada. Logo, realizou-se o ponto crítico dos cupons, montagem dos stumbs e metalização com ouro. A caracterização topográfica das superfícies contendo os biofilmes foi observada em microscópio eletrônico de varredura modelo VEGA 3 (TESCAN®), em aumentos de 2 kx, 10 kx e 15 kx.
2.5 TOLERÂNCIA ÀS CONDIÇÕES GÁSTRICAS E INTESTINAIS
A partir de culturas congeladas (n=18) em caldo Brain Heart Infusion (BHI) contendo 20% (v/v) de glicerol, foi retirada uma alíquota (10 μL) de cada isolado para ser inoculada em 5 mL de BHI e subcultivado a ٣٧°C durante 24h. Posteriormente, em triplicata, os isolados de Salmonella spp. foram submetidos a avaliação de tolerância as condições gástricas por meio de modelo in vitro, de acordo Santos et al.32 com modificações.
Para simular as condições gástricas, foi preparada uma solução de suco gástrico artificial no volume de 10 mL, contendo 6,2 g.L-1 NaCl, 2,2 g.L-1 KCl, 0,22 g.L-1 CaCl2 e 1,2 g.L-1 NaHCO3 e pH 2,5. As culturas a serem estudadas, foram ajustadas em 10 mL de solução salina esterilizada (0,85%) na escala 0,5 de McFarland, o que corresponde a 1,5 x 108 UFC.mL-1, e posteriormente, foi retirada uma alíquota de 6,0 mL de cada cultura e adicionada aos 10 mL da solução de suco gástrico artificial anteriormente preparado. Posteriormente, essa solução foi suplementada com 0,3% de pepsina (Sigma-Aldrich, St. Louis, MO, USA) e mantida a 37°C por uma hora, sob agitação orbital de 150 rpm em incubadora refrigerada (Thoth, Piracicaba, SP, Brasil). Após o período de incubação, uma alíquota de 1,0 mL foi removida para quantificação dos microrganismos presentes. Após diluição seriada, foi retirado o volume de 1,0 mL e inoculado em placas de petri e logo adicionado ágar Trypticase Soy Agar (TSA Oxoid®), as placas foram incubadas a 37°C durante 24h, sendo os resultados expressos em log10 UFC.mL-1.
Para simular a passagem através do intestino delgado, foram retirados 2,0 mL após as condições gástricas e transferidos para 8,0 mL de secreção duodenal artificial, a qual consistia em 6,4 g.L-1 NaHCO3, 0,239 g.L-1 KCl e 1,28 g.L-1 NaCl, pH 7,2, suplementada com 0,5% de sal biliar (Oxgall, Merck, Darmstadt, Germany) e 0,1% de pancreatina (Sigma-Aldrich, St Louis, MO, USA). Essa solução foi mantida a 37°C durante 3h sob agitação orbital de 150 rpm em incubadora refrigerada (Thoth, Piracicaba, SP, Brasil). Após o período de incubação, as contagens das células sobreviventes foram realizadas como descrito anteriormente na simulação das condições gástrica.
2.6 TAXA DE SOBREVIVÊNCIA
A taxa de microrganismos sobreviventes foi determinada de acordo com o descrito por Wang et al.33, usando a seguinte equação:
SR (%) = [log UFC N/ log UFC N0] * 100
Onde SR é a taxa de sobrevivência, N0 é a população de microrganismos antes do ensaio e N é a população de microrganismos depois do ensaio.
2.7 ANÁLISES ESTATÍSTICAS
Para análise estatística foi utilizado o Programa SPSS versão 19. Para determinar as diferenças estatísticas existentes entre as amostras, os resultados obtidos foram analisados pelo teste de Tukey-Kramer, com nível de significância de p<0,05, utilizando o programa XLSTAT.
3 RESULTADOS E DISCUSSÃO
Quando avaliada a formação de biofilme em superfície de polipropileno, todos os 13 isolados apresentaram essa capacidade, com contagens de células sésseis médias de 5,03 log UFC/cm2. Alguns genes específicos estão ligados a produção de fímbrias, adesão e produção de celulose, sendo lpfA, adrA, sipA e sdiA os principais. Assim, os 13 isolados apresentaram estes genes, comprovando de forma genotípica essa capacidade. As fimbrias desempenham um papel importante na síntese da matriz extracelular e ligação de microrganismos a superfície, favorecendo a adesão inicial, formação do biofilme e a perpetuação do microrganismo no ambiente34-35.
Aliado à estrutura de exopolissacarídeos do biofilme, microrganismos Gram-negativos, principalmente da família Enterobacteriaceae possuem uma membrana externa capaz de atuar como bloqueio à passagem de certos agentes36. As bactérias também possuem um sistema de efluxo, capaz de bombear substâncias como antibióticos e biocidas para fora da célula, tornando assim o agente utilizado ineficaz37-39. Essa resistência também foi observada com outros microrganismos que apresentam a capacidade de formação de biofilmes40-41.
Os biofilmes passam a ser um fator de virulência importante, pois favorecem a resistência dos microrganismos, facilitando a troca de material genético entre micro-organismos42. Os mecanismos que envolvem a formação do biofilme são complexos, sabe-se que a presença de genes relacionados a virulência e sensor de quórum (do inglês quorum sensing) são importantes (sipA e sdiA) pois favorecem a troca de informação entre os microrganismos e garantem sobrevivência sob condições adversas43-44.
Nosso estudo também comprovou a capacidade de formação de biofilme através de imagens de MEV, sendo possível avaliar a morfologia bacteriana e presença de exopolissacarídeos. A presença de EPS demonstra um fator de proteção bacteriana favorecendo sua adesão a superfície e sobrevivência em condições gastrointestinais.
Muitos genes de resistência a esses compostos podem estar em cassetes gênicos ligados a integrons, onde agregam-se ao DNA plasmidial e transferem a resistência para outras bactérias que habitam o mesmo ambiente45-46. Bactérias ESBL positivas, por apresentarem esta capacidade de transferência da resistência antimicrobiana através de plasmídeos, também podem favorecer a tolerância a biocidas47-49.
Os isolados de Salmonella spp. deste estudo apresentam o gene conservado em segmentos de integrons, que também inclui o gene de resistência ao antibiótico sulfonamida. Como este gene está presente em elementos genéticos móveis, favorece que a resistência se espalhe via transferência horizontal de genes, da mesma forma que pode contribuir para uma co-resistência a antibióticos e biocidas50-52.
Pelos dados da Tabela 3, foi observado que a quantificação de Salmonella spp. diminuiu (p<0,05) conforme a passagem do trato gastrointestinal, simulada através dos sucos gástricos e intestinais.
Após o contato com organismo humano, Salmonella spp. encontra várias barreiras de defesa do hospedeiro contra infecções. Desta forma, para que ocorra o desenvolvimento da doença, o microrganismo deve sobreviver em condições ácidas extremas dentro do estômago e concentrações elevadas de sais biliares. Além das baixas concentrações de oxigênio dentro do intestino e da competição com outros microrganismos que habitam o trato gastrintestinal53.
A dose infectante de Salmonella não tifóide é estimada em pelo menos 6 log6,54, no entanto, alguns fatores contribuem para que essa dose seja reduzida, visto que a acidez estomacal humana depende de variáveis fisiológicas como a ingestão alimentar, ansiedade, idade e medicamentos55. Assim, indivíduos afetados por hipocloridria (baixo nível de HCl no estômago) ou acloridria (falta de HCl no estômago) com diminuição ou perda do mecanismo de proteção do ácido gástrico19, por exemplo, terão uma dose infectante de Salmonella spp. inferior a indivíduos saudáveis. Essa dose também depende da fonte de alimento carreadora de Salmonella spp., que pode atuar como proteção à acidez estomacal, como alimentos ricos em gordura ou com elevado teor de proteína, por exemplo ovos e chocolate56.
A taxa de sobrevivência total média, para todos os isolados estudados foi de 88,3%, sendo as taxas individuais apresentadas na Tabela 4. Já a taxa de sobrevivência à fase gástrica simulada foi, em média, de 81,7%, ou seja, aproximadamente 1,5 x 108 UFC.mL-1 iniciais, cerca de 1,2 x 108 UFC.mL-1 sobreviveram às condições gástricas de baixo pH.
Essa alta taxa de sobrevivência pode ser associada aos mecanismos de adaptação desenvolvidos pelo patógeno para se adaptar aos desafios ambientais54. S. Typhimurium tem maior facilidade de sobrevivência em ambientes que apresentem pH neutro, no entanto possui uma variedade de mecanismos adaptativos que lhe permitem responder a desafios potencialmente letais, como estresses ácidos presentes no ambiente externo ou em um hospedeiro animal, como a resposta à tolerância ácida (ATR)19,54. Desse modo, os microrganismos podem responder ao ambiente ácido modificando enzimas para o controle do pH intracelular; induzindo a síntese de proteínas de choque ácido para prevenir ou reparar o dano macromolecular causado pelo estresse ácido; modificando a composição de sua membrana para manter um grau de fluidez adaptável à sua sobrevivência54. Esses mecanismos adaptativos desempenham um papel vital na sobrevivência desses organismos em alimentos ácidos ou durante a passagem pelo ácido estomacal19.
Para avaliar as mudanças na tolerância térmica e na capacidade de sobrevivência às condições gastrointestinais simuladas, Melo et al.57 utilizaram S. Enteritidis PT4 e S. Typhimurium PT4 inoculadas em carne de peito de frango e submeteram essas bactérias a seções sequenciais de estresse, observando que a exposição sequencial a múltiplas tensões sub-letais aumentou a tolerância térmica e a sobrevivência em condições gastrointestinais. Além disso, a temperatura também pode ser um fator para determinar a resistência a ácidos, onde as células de S. Enteritidis cultivadas a 37°C mostraram maior resistência ácida, do que aquelas cultivadas a 10°C58. Pode-se observar (Tabela 4) que, em relação à taxa de sobrevivência (SR) gástrica, a SR intestinal foi maior que 100% para todos os isolados estudados, podendo-se inferir que as condições intestinais simuladas in vitro não inibiram a multiplicação bacteriana.
Os ácidos biliares são moléculas anfipáticas de superfície atuando como um tipo de detergente, com potente atividade antimicrobiana, não suficiente, no entanto, para inibir o crescimento de bactérias entéricas, que desenvolveram mecanismos para resistir à ação da bile59-61. Alguns dos mecanismos de resistência mais estudados são o efluxo de sais biliares do citoplasma bacteriano diretamente para fora da célula; interações entre a bile e lipopolissacarídeos de membrana; e alteração da produção de proteínas em resposta a doses subletais de sais biliares, que aumentam os mecanismos de resistência das bactérias, preferencialmente quando o microrganismo está em fase logarítmica de crescimento59. Desse modo, pode-se considerar que a Salmonella spp. é tolerante aos sais biliares e capaz de sobreviver à exposição a fluidos gastrintestinais62.
Com os resultados obtidos na comparação de isolados produtores de ESBL e isolados não produtores de ESBL (Tabela 5), é possível inferir que, apesar da diferença estatística entre os isolados nas taxas de sobrevivência das fases gástrica e intestinal, não houve diferença (p>0,05) na taxa de sobrevivência total entre os isolados estudados.
Não se conhece exatamente as variáveis que possam ter influenciado a alta taxa de sobrevivência de Salmonella spp. que foi observada no presente estudo. Pesquisas indicam que o ácido gástrico é mais eficiente em eliminar bactérias na presença de nitrito salivar e ambos atuam sinergicamente para inativar os patógenos veiculados por alimentos antes da entrada destes no trato gastrointestinal19,55,63-64. Contudo, no presente estudo, o protocolo utilizado não utilizou o nitrito, assim como fizeram Santos et al.30.
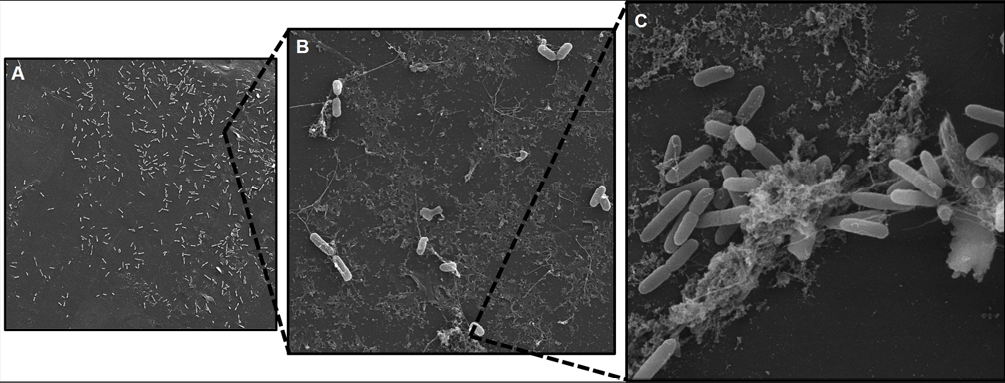
Figura 1 – Microscopia eletrônica de varredura (MEV) de biofilme de Salmonella Typhimurium (Isolado n˚ 1) em superfície de polipropileno com 96h/37 °C, em imagens de ٢ kx (A), 10 kx (B) e 15 kx (C).
Tabela 3 – Número de microrganismos sobreviventes (log UFC.mL-1) após a incubação sob condições gástricas e intestinais simuladas in vitro
|
Amostra |
Sorotipo |
População (log UFC.mL-1) |
|
|
Fase Gástrica |
Fase Intestinal |
||
|
1 |
S. Typhimurium |
7,15 ± 0,08a |
7,46 ± 0,23a |
|
2 |
S. Heidelberg |
7,14 ± 0,19b |
7,60 ± 0,22a |
|
3 |
S. Ndolo |
7,22 ± 0,06a |
7,69 ± 0,70a |
|
4 |
S. Heidelberg |
7,36 ± 0,03b |
7,84 ± 0,15a |
|
5 |
S. Heidelberg |
7,28 ± 0,10b |
8,11 ± 0,43a |
|
6 |
S. Ndolo |
7,20 ± 0,04b |
8,01 ± 0,28a |
|
7 |
S. Typhimurium |
7,27 ± 0,02b |
7,98 ± 0,34a |
|
8 |
S. Typhimurium |
7,26 ± 0,14b |
8,02 ± 0,11a |
|
9 |
S. Heidelberg |
7,45 ± 0,04b |
8,10 ± 0,35a |
|
10 |
S. Typhimurium |
7,17 ± 0,04b |
8,11 ± 0,18a |
|
11 |
S. Typhimurium |
7,33 ± 0,19b |
8,23 ± 0,25a |
|
12 |
S. Ndolo |
7,31 ± 0,16b |
8,29 ± 0,18a |
|
13 |
S. Typhimurium |
7,44 ± 0,16b |
8,09 ± 0,24a |
|
14 |
S. Heidelberg |
7,44 ± 0,06a |
7,70 ± 0,67a |
|
15 |
S. Heidelberg |
7,25 ± 0,08a |
7,67 ± 0,71a |
|
16 |
S. Heidelberg |
7,43 ± 0,10a |
7,86 ± 0,61a |
|
17 |
S. Typhimurium |
7,56 ± 0,14a |
7,82 ± 0,88a |
|
18 |
S. Typhimurium |
7,54 ± 0,16a |
7,70 ± 0,74a |
Nota: médias seguidas por letras iguais, na mesma linha, não diferem entre si pelo teste de Tukey a 5% de probabilidade. NC: 8,17 log UFC.mL-1.
Tabela 4 – Taxa de sobrevivência média dos microrganismos após a incubação sob condições gástricas e intestinais simuladas in vitro
|
Isolados |
Sorotipo |
Taxa de Sobrevivência (SR) em % |
||
|
SR Gástrica |
SR Intestinal |
SR total |
||
|
1 |
S. Typhimurium |
79,8b |
104,3a |
83,3b |
|
2 |
S. Heidelberg |
79,7b |
106,6a |
84,9b |
|
3 |
S. Ndolo |
80,7b |
106,5a |
85,9b |
|
4 |
S. Heidelberg |
82,2c |
106,6a |
87,6b |
|
5 |
S. Heidelberg |
81,3b |
111,4a |
90,6b |
|
6 |
S. Ndolo |
80,4c |
111,2a |
89,5b |
|
7 |
S. Typhimurium |
81,2b |
119,7a |
89,1b |
|
8 |
S. Typhimurium |
81,1c |
110,6a |
89,6b |
|
9 |
S. Heidelberg |
83,2b |
108,6a |
90,4b |
|
10 |
S. Typhimurium |
80,1c |
113,2a |
90,6b |
|
11 |
S. Typhimurium |
81,9c |
112,3a |
91,9b |
|
12 |
S. Ndolo |
81,7c |
113,4a |
92,6b |
|
13 |
S. Typhimurium |
83,1c |
108,7a |
90,3b |
|
14 |
S. Heidelberg |
83,1b |
103,4a |
86,0b |
|
15 |
S. Heidelberg |
80,9b |
105,8a |
86,7b |
|
16 |
S. Heidelberg |
82,9b |
105,7a |
87,7b |
|
17 |
S. Typhimurium |
84,4a |
103,3a |
87,3a |
|
18 |
S. Typhimurium |
84,2a |
102,0a |
86,0a |
Nota: médias seguidas por letras iguais, na mesma linha, não diferem entre si pelo teste de Tukey a 5% de probabilidade.
Tabela 5 – Taxa de sobrevivência média dos isolados produtores ou não produtores de enzimas beta-lactamases de espectro estendido (ESBL), após a incubação sob condições gástricas e intestinais simuladas in vitro
|
Isolados |
Taxa de Sobrevivência (SR) em % |
||
|
SR Gástrica |
SR Intestinal |
SR Total |
|
|
ESBL |
81,26b |
109,46a |
88,94a |
|
Não ESBL |
83,13a |
104,10b |
86,55a |
Nota: médias seguidas por letras iguais, na mesma coluna, não diferem entre si pelo teste de Tukey a 5% de probabilidade.
4 CONCLUSÃO
Os resultados deste estudo demonstram que, apesar da diminuição da contagem de microrganismos conforme a passagem simulada pelos sucos gástricos e intestinais, a taxa de sobrevivência média total dos isolados de Salmonella investigadas foi alta (88,3%), o que permite inferir que os isolados utilizados neste estudo possuem a capacidade de sobreviver às condições gastrointestinais simuladas, permanecendo em número capaz de causar infecção mesmo após a passagem por este ambiente hostil, de baixo pH, baixa concentração de oxigênio e presença de sais biliares. Essa alta taxa de sobrevivência pode estar relacionada a presença de mecanismos de resposta de Salmonella spp. às condições ácidas, que permitem a sobrevivência deste microrganismo em condições de estresse.
REFERÊNCIAS
- Majowicz SE, Musto J, Scallan E, Angulo FJ, Kirk M, O’Brien SJ, et al. The global burden of nontyphoidal Salmonella gastroenteritis. Clin Infect Dis. 2010;50(6):882-89.
- Scallan E, Hoekstra RM, Angulo FJ, Tauxe RV, Widdowson MA, Roy SL, et al. Foodborne illness acquired in the United States – major pathogens. Emerging Infect Dis. 2011;17(1):7.
- EFSA and ECDC – European Food Safety Authority and European Centre for Disease Prevention and Control. The European Union One Health 2018 Zoonoses Report. EFSA Journal. 2019;17(12):e05926.
- Brasil. Ministério da Saúde. Secretaria de Vigilância em Saúde. Departamento de Vigilância das Doenças Transmissíveis. Coordenação Geral de Doenças Transmissíveis. Surtos de doenças transmitidas por alimentos no Brasil: informe 2018. Brasília: Ministério da Saúde; 2019. [citado em 2023 Jun 5]. Disponível em: https://portalarquivos2.saude.gov.br/images/pdf/2019/fevereiro/15/Apresenta----o-Surtos-DTA--Fevereiro-2019.pdf
- Coburn B, Grassl GA, Finlay BB. Salmonella, the host and disease: a brief review. Immunol Cell Biol. 2007;85(2):112-18.
- Chen HM, Wang Y, Su LH, Chiu CH. Nontyphoid Salmonella infection: microbiology, clinical features, and antimicrobial therapy. Pediatr Neonatol. 2013;54(3):147-52.
- Cejas D, Vignoli R, Quinteros M, Marino R, Callejo R, Betancor L, et al. First detection of CMY-2 plasmid mediated β-lactamase in Salmonella Heidelberg in South America. Rev Argent Microbiol. 2014;46(1):30-33.
- Nadi ZR, Salehi TZ, Tamai IA, Sillanpaa M, Dallal MMS. Evaluation of antibiotic resistance and prevalence of common Salmonella enterica serovars isolated from foodborne outbreaks. Microchem Journal. 2020;155:104660.
- Wang H, Ye K, Wei X, Cao J, Xu X, Zhou G. Occurrence, antimicrobial resistance and biofilm formation of Salmonella isolates from a chicken slaughter plant in China. Food Control. 2013;33(2):378-84.
- Dar MA, Ahmad SM, Bhat SA, Ahmed R, Urwat U, Mumtaz PT, et al. Salmonella Typhimurium in poultry: a review. Worlds Poult Sci J. 2017;73(2):345-54.
- Voss-Rech D, Kramer B, Silva VS, Rebelatto R, Abreu PG, Coldebella A, et al. Longitudinal study reveals persistent environmental Salmonella Heidelberg in Brazilian broiler farms. Vet Microbiol. 2019; 233:118-23.
- Kottwitz LBM, Oliveira TCRM, Alcocer I, Farah SMSS, Abrahão WSM, Rodrigues DP. Avaliação epidemiológica de surtos de salmonelose ocorridos no período de 1999 a 2008 no Estado do Paraná, Brasil. Acta Sci Health Sci. 2010;32:9-15.
- Mendonça, EP. Características de virulência, resistência e diversidade genética de sorovares de Salmonella com impacto na saúde pública, isolados de frangos de corte no Brasil [tese de Doutorado]. Uberlândia: Universidade Federal de Uberlândia; 2016. 131 p.
- Perin AP, Martins BTF, Barreiros MAB, Yamatogi RS, Nero LA, Bersot LS. Occurrence, quantification, pulse types, and antimicrobial susceptibility of Salmonella sp. isolated from chicken meat in the state of Paraná, Brazil. Braz J Microbiol. 2019;51(1):335-45.
- Lejeune P. Biofilms – Dependent Regulation of Gene Expression. In: Wilson M, Devine D, organizators. Medical Implications of Biofilms. Cambridge: Cambridge University Press; 2003. p. 3-17.
- Oliveira DCV. Produção de biofilme por Salmonella sp. isolada de frango [dissertação de mestrado]. Botucatu: Instituto de Biociências de Botucatu, Universidade Estadual Paulista; 2011. 75 p.
- Carattoli A. Plasmids and the spread of resistance. Int J Med Microbiol. 2013;303(6-7):298-304.
- Sirsat SA, Burkholder KM, Muthaiyan A, Dowd SE, Bhunia AK, Ricke SC. Effect of sublethal heat stress on Salmonella Typhimurium virulence. J Appl Microbiol. 2011;110(3):813-22.
- Smith JL. The role of gastric acid in preventing foodborne disease and how bacteria overcome acid conditions. J Food Prot. 2003;66(7):1292-1303.
- Berk PA, De Jonge R, Zwietering MH, Abee T, Kieboom, J. Acid resistance variability among isolates of Salmonella enterica serovar Typhimurium DT104. J Appl Microbiol. 2005;99(4):859-866.
- Howden CW, Hunt RH. Relationship between gastric secretion and infection. Gut. 1987;28(1):96.
- McDonald SW, MacFarlane NG. The mouth, stomach and intestines. Anaesth Intensive Care Med. 2018;19(3):128-32.
- Crum-Cianflone NF. Salmonellosis and the gastrointestinal tract: more than just peanut butter. Curr Gastroenterol Rep. 2008;10(4):424-31.
- Tadielo LE, Bellé TH, Santos EAR, Schmiedt JA, Cerqueira-Cézar CK, Nero LA, et al. Pure and mixed biofilms formation of Listeria monocytogenes and Salmonella Typhimurium on polypropylene surfaces. LWT – Food Sci and Technol. 2022;162:113469.
- Poimenidou SV, Chrysadakou M, Tzakoniati A, Bikouli VC, Nychas GJ, Skandamis PN. Variability of Listeria monocytogenes strains in biofilm formation on stainless steel and polystyrene materials and resistance to peracetic acid and quaternary ammonium compounds. Int J Food Microbiol. 2016;237:164-71.
- Dias RC, Santos BC, Santos LF, Vieira MA, Yamatogi, RS, Mondelli AL, et al. Diarrheagenic Escherichia coli pathotypes investigation revealed atypical enteropathogenic E. coli as putative emerging diarrheal agents in children living in Botucatu, São Paulo State, Brazil. APMIS. 2016;124(4):299–308.
- Oliveira DCV, Fernandes Júnior A, Kaneno R, Silva MG, Araújo Júnior JP, Silva NCC, et al. Ability of Salmonella spp. to Produce Biofilm Is Dependent on Temperature and Surface Material. Foodborne Pathog Dis. 2014;11(6):478-83.
- Bäumler AJ, Heffron F. Identification and sequence analysis of lpfABCDE, a putative fimbrial operon of Salmonella Typhimurium. J. Bacteriol. 1995;177(8):2087-97.
- Collinson SK, Emody L, Trust TJ, Kay WW. Thin aggregative fimbriae from diarrheagenic Escherichia coli. J. Bacteriol. 1992;174(13):4490-95.
- Halatsi K, Oikonomou I, Lambiri M, Mandilara G, Vatopoulos A, Kyriacou A. PCR detection of Salmonella spp. using primers targeting the quorum sensing gene sdiA. FEMS Microbiol Lett. 2006;259(2):201-07.
- Skyberg JA, Logue CM, Nolan LK. Virulence genotyping of Salmonella spp. with multiplex PCR. Avian Dis. 2006;50(1):77-81.
- Santos KMO, Vieira ADS, Buriti FCA, Nascimento JCF, Melo MES, Bruno LM, et al. Artisanal coalho cheeses as source of beneficial Lactobacillus plantarum and Lactobacillus rhamnosus strains. Dairy Sci Technol. 2015;95(2):209-230.
- Wang H, Ye K, Wei X, Cao J, Xu X, Zhou G. Occurrence, antimicrobial resistance and biofilm formation of Salmonella isolates from a chicken slaughter plant in China. Food Control. 2013;33(2):378-384.
- Barnhart MM, Chapman MR. Curli biogenesis and function. Annu Ver Microbiol. 2006;60:131-47.
- Borsoi A, Santin E, Santos LR, Salle CTP, Moraes HLS, Nascimento VP. Inoculation of newly hatched broiler chicks with two Brazilian isolates of Salmonella Heidelberg strains with different virulence gene profiles, antimicrobial resistance, and pulsed field gel electrophoresis patterns to intestinal changes evaluation. Poult Sci. 2009;88(4):750-58.
- Morente EO, Fernández-Fuentes MA, Burgos MJG, Abriouel H, Pulido RP, Gálvez A. Biocide tolerance in bacteria. Int J Food Microbiol. 2013;162(1),13-25.
- Poole K. Efflux pumps as antimicrobial resistance mechanisms. Ann Med. 2007;39(3):162-76.
- Jiang X, Yu T, Liang Y, Ji S, Guo X, Ma J, et al. Efflux pump-mediated benzalkonium chloride resistance in Listeria monocytogenes isolated from retail food. Int J Food Microbiol. 2016;217:141-45.
- Weston N, Sharma P, Ricci V, Piddock LJV. Regulation of the AcrAB-TolC efflux pump in Enterobacteriaceae. Res Microbiol. 2018;169(7-8):425-31.
- Lee SHI, Cappato LP, Corassin CH, Cruz AGD, Oliveira CAFD. Effect of peracetic acid on biofilms formed by Staphylococcus aureus and Listeria monocytogenes isolated from dairy plants. J Dairy Sci. 2016;99(3):2384-90.
- Akinbobola AB, Sherry L, Mckay WG, Ramage G, Williams C. Tolerance of Pseudomonas aeruginosa in in-vitro biofilms to high-level peracetic acid disinfection. J Hosp Infec. 2017;97(2):162-68.
- Haubert L, Zehetmeyr ML, Pereira YMN, Kroning IS, Maia DSV, Sehn CP, et al. Tolerance to benzalkonium chloride and antimicrobial activity of Butia odorata Barb. Rodr. extract in Salmonella spp. isolates from food and food environments. Food Res Int. 2019;116:652-59.
- Bai AJ, Rai VR. Bacterial quorum sensing and food industry. Compr Rev Food Sci Food Saf. 2011;10(3):183-93.
- Hiller CC, Lucca V, Carvalho D, Borsoi A, Borges KA, Furian TQ, et al. Influence of catecholamines on biofilm formation by Salmonella Enteritidis. Microb Pathog. 2019;130:54-58.
- Cantón R, Ruiz-Garbajosa P. Co-resistance: an opportunity for the bacteria and resistance genes. Curr Opin Pharmacol. 2011;11(5):477-85.
- Paul D, Chakraborty R, Mandal SM. Biocides and health-care agents are more than just antibiotics: Inducing cross to co-resistance in microbes. Ecotoxicol Environ Saf. 2019;174:601-10.
- Silva N, Carvalho I, Currie C, Sousa M, Igrejas G, Poeta P. Extended‐Spectrum‐β‐Lactamase and Carbapenemase‐Producing Enterobacteriaceae in Food‐Producing Animals in Europe: An Impact on Public Health?. In: Capelo-Martínez JL, Igrejas G, organizators. Antibiotic Drug Resistence. Hoboken: John Wiley & Sons; 2019. p. 261-73.
- Poole TL, Callaway TR, Norman KN, Scott HM, Loneragan GH, Ison SA, et al. Transferability of antimicrobial resistance from multidrug-resistant Escherichia coli isolated from cattle in the USA to E. coli and Salmonella Newport recipients. J Glob Antimicrob Resist. 2017;11:123-32.
- Harakeh S, Azhar E, Almasaudi S, Kissoyan KB, Fadlallah S, Tanelian A, et al. Effects of a specific nutrient combination on ESBL resistance. Saudi J Biol Sci. 2019;26(7):1576-80.
- Kücken D, Feucht HH, Kaulfers PM. Association of qacE and qacE Δ1 with multiple resistance to antibiotics and antiseptics in clinical isolates of Gram-negative bacteria. FEMS Microbiol Lett. 2000;183(1):95-98.
- Chapman JS. Biocide resistance mechanisms. Int Biodeterior Biodegrad. 2003;51(2):133-38.
- Long M, Lai H, Deng W, Zhou K, Li B, Liu S, et al. Disinfectant susceptibility of different Salmonella serotypes isolated from chicken and egg production chains. J Appl Microbiol. 2016;121(3):672-81.
- Shah J, Desai PT, Chen D, Stevens JR, Weimer BC. Preadaptation to cold stress in Salmonella enterica serovar Typhimurium increases survival during subsequent acid stress exposure. Appl Environ Microbiol. 2013;79(23):7281-89.
- Álvarez-Ordóñez A, Begley M, Prieto M, Messens W, López M, Bernardo A, et al. Salmonella spp. survival strategies within the host gastrointestinal tract. Microbiol. 2011;157(12):3268-81.
- Dykhuizen RS, Frazer R, Duncan C, Smith CC, Golden M, Benjamin N, et al. Antimicrobial effect of acidified nitrite on gut pathogens: importance of dietary nitrate in host defense. Antimicrob Agents Chemother. 1996;40(6):1422-25.
- Mikkelsen LL, Naughton PJ, Hedemann MS, Jensen BB. Effects of physical properties of feed on microbial ecology and survival of Salmonella enterica serovar Typhimurium in the pig gastrointestinal tract. Appl Environ Microbiol. 2004;70(6):3485-92.
- Melo ANF, Souza GT, Schaffne D, Oliveira TCM, Maciel JF, Souza EL, et al. Changes in thermo-tolerance and survival under simulated gastrointestinal conditions of Salmonella Enteritidis PT4 and Salmonella Typhimurium PT4 in chicken breast meat after exposure to sequential stresses. Int J Food Microbiol. 2017;251:15-23.
- Yang Y, Khoo WJ, Zheng Q, Chung HJ, Yuk HG. Growth temperature alters Salmonella Enteritidis heat/acid resistance, membrane lipid composition and stress/virulence related gene expression. Int J Food Microbiol. 2014;172:102-109.
- Gunn JS. Mechanisms of bacterial resistance and response to bile. Microbes Infect. 2000;2(8):907-13.
- Merritt ME, Donaldson JR. Effect of bile salts on the DNA and membrane integrity of enteric bacteria. J Med Microbiol. 2009;58(12):1533-41.
- Pradhan D, Negi VD. Stress-induced adaptations in Salmonella: a ground for shaping its Pathogenesis. Microbiological research. 2019;229:126311.
- Van Velkinburgh JC, Gunn JS. PhoP-PhoQ-regulated loci are required for enhanced bile resistance in Salmonella spp. Infect and Immun. 1999;67(4):1614-22.
- McKnight, GM, Duncan CW, Liefert C, Golden MH. Dietary nitrate in man: friend or foe?. Br J Nutr. 1999;81:349-58.
- Xu J, Xu X, Verstraete W. The bactericidal effect and chemical reactions of acidified nitrite under conditions simulating the stomach. J Appl Microbiol. 2001;90(4):523-529.
* Mestre e doutoranda em Ciência Animal pela Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
† Mestre em Ciência Animal pela Universidade Federal do Paraná (UFPR) e doutoranda em Medicina Veterinária pela Universidade Estadual Paulista (UNESP), campus Botucatu, Botucatu, São Paulo, Brasil.
‡ Mestre em Ciência Animal pela Universidade Federal do Paraná (UFPR) e doutorando em Medicina Veterinária pela Universidade Estadual Paulista (UNESP), campus Botucatu, Botucatu, São Paulo, Brasil.
§ Pós-doutorado em Bovinocultura Leiteira pela Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
¶ Médica Veterinária pela Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
** Mestre em Ciência Animal pela Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
†† Doutor em Ciência e Tecnologia dos Alimentos pela Universidade Federal de Pelotas (UFPel). Docente na Universidade Estadual Paulista (Unesp), campus Botucatu, Botucatu, São Paulo, Brasil.
‡‡ Doutora em Medicina Veterinária pela Universidade Estadual Paulista (Unesp), campus Botucatu, Botucatu, São Paulo, Brasil.
§§ Mestre em Ciência Animal pela Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
¶¶ Médico Veterinário pela Universidade Federal de Pelotas (UFPel). Mestrando em Ciência Animal pela Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
*** Doutor em Ciência de Alimentos pela Universidade Estadual de Maringá (UEM). Docente na Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.
††† Doutor em Ciência dos Alimentos pela Universidade de São Paulo (USP). Docente na Universidade Federal do Paraná (UFPR), campus Palotina, Palotina, PR, Brasil.