
Artigo original
Padronização da Técnica RT-qPCR para detecção de Arbovírus em amostras de tecidos parafinados
Standardization of RT-qPCR Technique for Arbovirus detection in paraffin-embedded tissue samples
Maria Clara Valente de Freitas* Karla Fabiane Lopes de Melo†
Karla Fabiane Lopes de Melo† Edna Cristina Franco‡
Edna Cristina Franco‡ Samir Mansour Moraes Casseb§
Samir Mansour Moraes Casseb§

https://doi.org/10.18593/evid.32787
Recebido em 01 de junho de 2023 | Aceito em 18 de outubro de 2023
Resumo:
Arbovírus são transmitidos por mosquitos e carrapatos, causando doenças graves em humanos, como dengue, Zika, Chikungunya e febre amarela. Este estudo buscou padronizar a técnica de RT-qPCR para detecção desses vírus em amostras de tecidos parafinados. Foram utilizadas 45 amostras parafinadas, incluindo arbovírus em estudo e amostras de controle negativo, provenientes de diferentes períodos e tecidos, catalogadas pelo Instituto Evandro Chagas. A padronização da técnica de RT-qPCR é essencial para a vigilância epidemiológica de arbovírus na região amazônica e no Brasil. Os resultados demonstraram eficiência na detecção e quantificação do genoma viral dos diferentes arbovírus. A técnica de RT-qPCR se mostrou uma ferramenta importante para o diagnóstico diferencial em amostras de tecidos parafinados. Essa padronização é relevante para o monitoramento e controle efetivo das doenças transmitidas por arbovírus, contribuindo para a saúde pública e a prevenção de surtos. Este estudo ressalta a importância da padronização da técnica de RT-qPCR para detecção de arbovírus em amostras de tecidos parafinados. A região amazônica e o Brasil são áreas propensas a surtos dessas doenças, tornando esses avanços ainda mais significativos. A detecção precoce e precisa dos arbovírus permite a implementação de medidas de controle e prevenção mais eficazes, além de contribuir para a compreensão da epidemiologia dessas doenças.
Palavras-chave: Arbovírus. RT-qPCR. Tecidos Parafinados. Vigilância Epidemiológica. Diagnóstico Diferencial.
Abstract:
Arboviruses are transmitted by mosquitoes and ticks, causing severe diseases in humans such as dengue, Zika, Chikungunya, and yellow fever. This study aimed to standardize the RT-qPCR technique for detecting these viruses in paraffin-embedded tissue samples. A total of 45 paraffin-embedded samples were used, including arboviruses under study and negative control samples from different periods and tissues, cataloged by the Evandro Chagas Institute. Standardization of the RT-qPCR technique is essential for arbovirus surveillance in the Amazon region and Brazil. The results demonstrated efficiency in detecting and quantifying the viral genome of different arboviruses. The RT-qPCR technique proved to be an important tool for the differential diagnosis in paraffin-embedded tissue samples. This standardization is relevant for monitoring and effectively controlling arbovirus-transmitted diseases, contributing to public health and outbreak prevention. This study highlights the importance of standardizing the RT-qPCR technique for detecting arboviruses in paraffin-embedded tissue samples. The Amazon region and Brazil are prone to outbreaks of these diseases, making these advancements even more significant. Early and accurate detection of arboviruses enables the implementation of more effective control and prevention measures, as well as contributing to the understanding of the epidemiology of these diseases.
Keywords: Arboviruses. RT-qPCR. Paraffin-embedded Tissues. Epidemiological Surveillance. Differential Diagnosis.
@Autor correspondente: Doutor em Virologia; Instituto Evandro Chagas, Ananindeua, Pará; Universidade Federal do Pará (UFPA), Belém, Pará, Brasil; Instituto Evandro Chagas, Ananindeua, Pará; https://orcid.org/0000-0002-7419-3381; samircasseb@ufpa.br.
1 INTRODUÇÃO
Os arbovírus são vírus transmitidos por artrópodes, como mosquitos e carrapatos, e são responsáveis por várias doenças graves em humanos. Entre os arbovírus mais conhecidos e estudados, destacam-se os vírus da dengue (VDEN), zika (VZIK), chikungunya (VCHIK) e febre amarela (VFA). Cada um desses vírus possui características únicas, mas compartilham semelhanças em relação à transmissão, prevenção e controle1.
O VDEN é um dos arbovírus mais comuns e amplamente disseminados no mundo. É transmitido principalmente pelo mosquito Aedes aegypti, que se reproduz em áreas urbanas e se prolifera em recipientes de água parada. A dengue pode causar desde sintomas leves, como febre, dores de cabeça e no corpo, até formas graves que podem levar à hospitalização e até mesmo à morte. A prevenção da dengue envolve o combate ao mosquito vetor, eliminando os criadouros e utilizando medidas de proteção individual, como o uso de repelentes e telas em janelas e portas2.
O VDEN possui quatro sorotipos distintos, mas antigenicamente relacionados (VDEN1, VDEN2, VDEN3 e VDEN4). Os sorotipos de VDEN possuem 67-73% identidade nucleotídica (nt) e 69-78% de identidade ao nível de aminoácidos (aa). De três a seis subtipos (genótipos) podem ser distinguidos em cada sorotipo. O vírus Zika ganhou destaque mundial durante o surto ocorrido entre 2015 e 20162-3.
O Aedes aegypti também é o principal vetor de transmissão desse vírus, mas a transmissão sexual também foi relatada. O Zika pode causar sintomas semelhantes aos da dengue, mas o aspecto mais preocupante é a associação com a microcefalia em bebês nascidos de mães infectadas durante a gestação. Além disso, o Zika também pode estar relacionado a complicações neurológicas, como a síndrome de Guillain-Barré. A prevenção do Zika envolve as mesmas medidas de controle do mosquito vetor da dengue3.
A Chikungunya é outra doença viral transmitida pelo Aedes aegypti, assim como pelo Aedes albopictus. Os sintomas incluem febre alta, dores articulares intensas e exantema. A doença pode se tornar crônica e levar a problemas articulares de longo prazo. O controle da Chikungunya também envolve a eliminação de criadouros do mosquito vetor e o uso de medidas individuais de proteção4.
A febre amarela é transmitida principalmente pelos mosquitos Haemagogus e Sabethes em áreas silvestres, e pelo Aedes aegypti em áreas urbanas. A doença pode variar desde formas leves até formas graves, com manifestações hemorrágicas e falência de múltiplos órgãos. A vacinação é a principal forma de prevenção da febre amarela, além do controle do vetor em áreas urbanas5.
Em comum, todos esses arbovírus apresentam um impacto significativo na saúde pública, com potencial para surtos e epidemias. O controle eficaz dessas doenças envolve medidas de prevenção que visam principalmente a eliminação de criadouros do mosquito vetor, além da pesquisa científica contínua para desenvolvimento de vacinas e tratamentos eficazes6-7.
Diante da globalização e consequente aumento da urbanização, viagens internacionais e das mudanças climáticas vêm aumentando a exposição humana a novos e velhos Arbovírus. Deste modo, as infecções virais transmitidas por mosquitos vem se intensificando em regiões endêmicas e também em áreas que historicamente não são expostas a esses vírus8.
Desta maneira se torna cada vez mais cruciais para áreas como a região amazônica o diagnóstico diferencial para arbovírus, principalmente os citados acima, por métodos moleculares. Este problema acentua-se quando se utilizam amostras oriundas de blocos parafinados, pois métodos padronizados para este tipo de amostra são difíceis e poucos laboratórios se disponibilizam a realizar esta padronização dentro da rede pública de saúde.
Assim, a padronização de métodos moleculares como a técnica de Reação em Cadeia da Polimerase em Tempo Real (RT-qPCR) para amostras parafinadas torna-se de grande importância para o monitoramento do vírus dengue, tanto na área amazônica quanto no território brasileiro, dentro da vigilância epidemiológica.
2 MATERIAIS E MÉTODOS
2.1 AMOSTRAS PARAFINADAS
As amostras parafinadas utilizadas neste estudo estão todas armazenadas e catalogadas na Seção de Patologia (SAPAT) do Instituto Evandro Chagas. Onde cada uma delas possui número de registro e ficha epidemiológica com a suspeita clínica e resultado realizado por imuno-histoquímica (IHQ) e Teste Histopatológico (HE) aprovadas pelo Comitê de Ética em Pesquisa – CEP de acordo com o número do parecer 4.909.596 em conformidade com a Resolução do Conselho Nacional de Saúde/Ministério da Saúde nº 466/12 Publicada no DOU nº 12 no dia 13 de junho de 2013.
Foram utilizadas 45 amostras de diferentes períodos e tecidos, sendo a seguinte distribuição: 10 casos confirmados de Febre Amarela;10 casos confirmados de Chikungunya; 10 casos confirmados de Dengue; 10 casos confirmados de Zika e 5 casos negativos para qualquer um dos arbovírus citados.
2.2 ESTOQUE VIRAL
Para o estoque viral foram utilizados uma cepa de cada um dos seguintes vírus:
Tabela 1 – Estoque viral (unidade formadora de placa (pfu)/ml)
|
Amostra |
Vírus |
PFU/ml |
|
ISO_DEN01 |
Dengue 1 (VDEN1) |
3,6 x 106 |
|
ISO_DEN02 |
Dengue 2 (VDEN2) |
7,8x107 |
|
ISO_DEN03 |
Dengue 3 (VDEN3) |
8,5x106 |
|
ISO_DEN04 |
Dengue 4 (VDEN4) |
1,3x106 |
|
ISO_ZIK |
Zika (VZIK) |
8,3x108 |
|
ISO_CHIK |
Chikungunya (VCHIK) |
2,9x106 |
|
ISO_FA |
Febre Amarela (VFA) |
1,4x106 |
Cada cepa viral foi inoculada em um frasco de cultura de 75 cm2 de área, contendo uma monocamada de células VERO, cultivadas em 10 mL de meio DMEM, suplementado com 5% de SBF e antibióticos/antifúngico. Primeiramente, o meio de cultura DMEM foi aspirado do frasco contendo a monocamada celular,que foi lavada 3X com PBS; após isso inoculado 1 mL da suspensão viral e incubada durante 1 h, a 37°C, em estufa de CO2, para permitir a adsorção e penetração viral e, então, o inóculo foi retirado por sucção a vácuo e 10 mL do meio DMEM com 5% de Soro Bovino Fetal (SBF). Foram observadas as garrafas por cerca de 5 dias após a infecção (dpi). Sendo que o sobrenadante de cada garrafa foi retirado, após se observar citopático viral (ECP) de cerca de 70% da monocamada ou até 5 dpi. Estas alíquotas foram congeladas a –80°C até o momento de uso.
2.3 DETERMINAÇÃO DO TÍTULO INFECCIOSO VIRAL
Para o título infeccioso do estoque viral e das doses infectantes foi utilizada a técnica de TCID50, conforme descrito por Reed e Muench9. Nesta técnica, a partir de uma suspensão-viral foi preparada uma série de diluição na razão de 1:10, em tubos de microtitulação, com meio DMEM sem SBF (225 µL de meio DMEM + 25 µL de suspensão viral). Após, 200 µL destas diluições foram adicionadas ao tapete celular (densidade celular 2x105 células/mL) previamente formado em placas de 96 cavidades, após aspiração do meio de cultura (5% de SBF + antibiótico/antifúngico). A placa então, foi incubada a 37°C, em estufa de CO2, durante 48 h e observada em microscópio invertido. Caso a diluição que infecta 50% das cavidades variar entre duas diluições, o título pode ser calculado através da distância proporcional (DP) entre as mesmas, pelas seguintes fórmulas:
DP= (% de positivas acima de 50%) – 50% / (% de positivas acima de 50%) – (% de positivas abaixo de 50%) e log TCID50 = (DP x log do fator de diluição) + (log da diluição acima de 50%). Sendo que o valor obtido será na unidade TCID50/mL.
2.4 OBTENÇÃO DO RNA VIRAL
O RNA viral do estoque viral e já determinado o título infeccioso foram extraídos utilizando o kit comercial ReliaPrep™ Viral TNA Miniprep System (Promega, EUA) de acordo com as especificações do fabricante. Para as amostras de parafina foi utilizado o produto GenElute™-E FFPE Deparaffinization Solution (MERCK, ALE) para melhorar a retirada da parafina do material biológico e em seguida utilizado o kit comercial ReliaPrep™ FFPE RNA Total (Promega, EUA) de acordo com as instruções do fabricante. A determinação da concentração e pureza do dos RNAs das duas formas de extração foi determinada pela plataforma Qubit 4 (Thermo Fisher Scientific, EUA), utilizando o kit comercial Qubit RNA Assay (Invitrogen, EUA) conforme descrição do fabricante, e pela plataforma 2100 Bioanalyzer (Agilent Technologies, ALE), utilizando o kit comercial Pico 6000 RNA (Agilent Technologies, ALE) conforme descrito pelo fabricante, respectivamente.
2.5 DETECÇÃO E QUANTIFICAÇÃO DO GENOMA VIRAL POR RT-QPCR
Para esta etapa, foi utilizada a plataforma 7500 Real-Time PCR System (Thermo Scientific, EUA). Sendo que para detecção e quantificação da carga viral foi utilizado o método descrito por Johnson et al.10 para os sorotipos de VDEN, VZIK, VFA e VCHIK; Além destes iniciadores, foi utilizado um par de iniciadores para controle endógeno vendido comercialmente B-100 (Qiagen, ALE). É importante ressaltar que todos estes protocolos foram utilizados para o diagnóstico de acordo com o Ministério da Saúde (MS).
Todos os protocolos descritos utilizaram o kit comercial GoTaq Probe 1-Step RT-qPCR System (Promega, EUA). Em conjunto com os métodos relatados acima foi realizada a quantificação de carga viral como escrito por Gustafsson et al.11 e Jonsson et al.12, onde foram utilizadas amostras quantificadas pelo método de TCID50 para determinar o número de cópias/mL de cada estoque viral. Importante ressaltar que cada amostra foi realizada em triplicata técnica, menos os controles para quantificação que foram feitos cada diluição pelo menos 10 vezes para aumentar a confiabilidade da diluição.
2.6 ESPECIFICIDADE DO RT-QPCR
Para esta etapa foi utilizado o painel de amostras parafinadas positivas para cada um dos arbovírus, sendo que cada amostras que foi submetida ao RT-qPCR possui resultado positivo para imunohistoquímica (IHQ) e teste histopatológico com hematoxilina/eosina (HE) assim sendo possível realizar a comparação com os resultados obtido por RT-qPCR.
2.7 ANÁLISE ESTATÍSTICA
Para análises estatísticas, foram utilizados o programa R-project (r-project.org/) e o programa Jamovi v 2.3 (jamovi.org), sendo implementado os seguintes teste para as análises dos resultados: ANOVA, t-student, no entanto antes de cada análise foi realizado um teste de normalidade dos dados. Todos estes testes foram avaliados e considerados significante com p-valor<0,05.
3 RESULTADOS
Inicialmente foi realizado o teste para avaliar a sensibilidade e eficiência para os iniciadores utilizados neste trabalho. Para isto utilizamos amostras que foram infectadas em células vero e quantificadas pelo método de TCID50.
É possível observar que no teste realizado no VDEN (Fig. 1a), os iniciadores foram detectáveis de 1x108 até 1x101 PFU/mL demonstrando uma alta capacidade de detecção. E, concomitantemente, o teste realizado com VZIK (Fig. 1b), teve sua eficiência bem semelhante ao visto em VDEN (Fig. 1a). No teste realizado com VFA (Fig. 1c), observou-se uma pequena diferença dos resultados dos testes realizados com VDEN e VZIK. Onde os valores de Cq foram inferiores aos anteriores resultando uma maior sensibilidade deste conjunto de iniciadores em relação aos de VZIK e VDEN. Ao realizar o teste de eficiência para VCHIK (Fig. 1d), demonstra-se que o mesmo apenas consegue amplificar de forma aceitável valores acima de 1x102 PFU/mL mostrando que este conjunto de iniciadores é o menos sensível de todos os testados.
Vale ressaltar que apesar de pequenas variações todos os iniciadores demonstraram-se eficazes e com uma grande capacidade eficiência e sensibilidade. Podemos observar isso na Tabela 2.
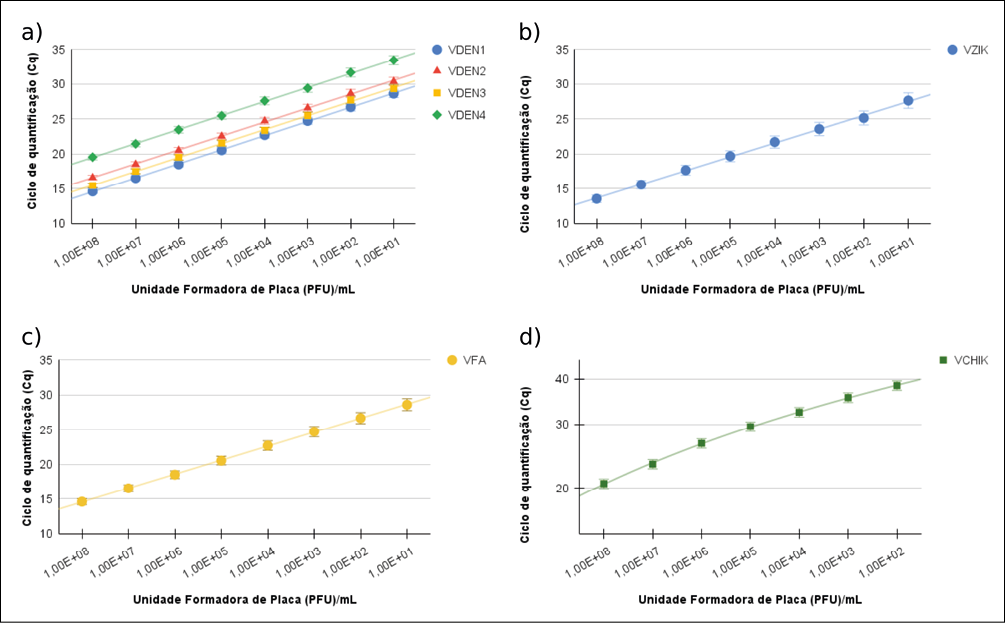
Figura 1 – Curva de eficiência de RT-qPCR para o vírus da dengue (VDEN) 1-4 (a), vírus da Zika (VZIK) (b), vírus da febre amarela (VFA) (c), e vírus Chikungunya (VCHIK) (d). Unidade formadora de placa (PFU)/mL por ciclo de quantificação (Cq).
Tabela 2 – Tabela com valores de R2 e Eficiência em %
|
VDEN1 |
VDEN2 |
VDEN3 |
VDEN4 |
VZIK |
VFA |
VCHIK |
|
|
Y |
37.037 |
39,496 |
38.594 |
37.192 |
38.476 |
38.109 |
39.006 |
|
R2 |
0,996 |
0,999 |
1 |
0,999 |
1 |
0,997 |
0,999 |
|
Eficiência (%) |
95.385 |
94.885 |
95.245 |
95.518 |
95.672 |
95.379 |
92.853 |
4 DISCUSSÃO
Diante da globalização e consequente aumento da urbanização, viagens internacionais e das mudanças climáticas vêm aumentando a exposição humana a novos e velhos Arbovírus8. Dessa forma, torna-se cada vez mais importante para áreas endêmicas, como a região amazônica, o diagnóstico individualizado para arbovírus, principalmente os que foram analisados neste estudo, por meio de métodos moleculares.
A técnica de RT-qPCR tem sido amplamente utilizada para a detecção e diagnóstico de diversos agentes infecciosos, incluindo os arbovírus. No contexto específico de amostras de parafina, a aplicação da RT-qPCR para a detecção de arbovírus apresenta desafios únicos, mas também oferece benefícios significativos13.
As amostras de parafina são amplamente utilizadas na preservação de tecidos e células para análises histopatológicas e diagnóstico de doenças. Um dos principais benefícios do uso da RT-qPCR em amostras de parafina é a capacidade de realizar análises retrospectivas. Isso significa que amostras antigas, que foram preservadas em blocos de parafina por longos períodos de tempo, podem ser recuperadas e testadas para a presença de arbovírus. Isso é particularmente útil em estudos epidemiológicos, permitindo uma compreensão mais abrangente da distribuição temporal e geográfica desses vírus14.
A detecção de Dengue em amostras de parafina utilizando a técnica de RT-qPCR tem sido objeto de estudos científicos. Estudo realizado por Guerra et al.13 demonstrou a detecção bem-sucedida do VDEN em amostras de fígado preservadas em parafina. No entanto, os autores não foram capazes de realizar a sorotipagem de dengue das amostras oriundas dos blocos de parafina.
Os resultados obtidos em nosso estudo para amostras de VDEN foram consistentes com os resultados encontrados no estudo de Guerra et al.13 em eficiência e capacidade de detecção. No entanto, nossos resultados apresentam uma contribuição adicional significativa, pois além de detectar o genoma viral, também conseguimos identificar o sorotipo específico responsável pela infecção. Esse aspecto é de extrema importância, especialmente para estudos epidemiológicos, uma vez que fornece informações adicionais não apenas para a detecção, mas também para uma melhor compreensão dos sorotipos que estão circulando na população.
A capacidade de identificar o sorotipo viral é de grande valia, pois cada sorotipo do VDEN pode apresentar características clínicas e epidemiológicas distintas. Portanto, poder distinguir entre os diferentes sorotipos em circulação ajuda a elucidar padrões de propagação do vírus e a identificar possíveis surtos ou tendências epidemiológicas. Além disso, essa informação auxilia na implementação de medidas de controle e prevenção mais efetivas.
Ao considerar a detecção e identificação dos sorotipos do VDEN em amostras de parafina, estamos ampliando o conhecimento sobre a epidemiologia dessa doença. Isso nos permite compreender melhor a dinâmica de transmissão do vírus, os padrões sazonais e geográficos de infecção, bem como a resposta imunológica da população.
Em nosso estudo, também investigamos a detecção do VZIK em amostras provenientes de tecidos parafinados. Vale ressaltar que a eficácia da técnica de RT-qPCR na detecção retrospectiva do VZIK em amostras de parafina já foi demonstrada, permitindo a investigação de casos clínicos passados e estudos epidemiológicos14.
Nossos resultados foram consistentes com o estudo de Reagan-Steiner et al.15, que analisou a presença do VZIK em tecidos placentários e fetais, tanto parafinados quanto não parafinados. Observamos uma alta concordância com os achados descritos na literatura, o que confirma a eficiência da nossa abordagem na detecção do VZIK em amostras de parafina. Essa consistência fortalece a validade e a confiabilidade da nossa técnica, contribuindo para o avanço do conhecimento sobre a infecção pelo VZIK e facilitando o desenvolvimento de estratégias mais eficazes de prevenção e controle.
Em nosso estudo, conduzimos testes para a detecção do VFA utilizando amostras vacinais e amostras selvagens. É importante destacar que nossa metodologia foi desenvolvida de forma a possibilitar a detecção de qualquer uma das cepas virais relacionadas à febre amarela.
Nossos resultados revelaram um alto índice de eficiência e especificidade, superando os 95%, o que é muito promissor para a detecção do VFA em diferentes tipos de amostras, incluindo amostras vacinais e selvagens.
O estudo de Domingo et al.16 também corroborou nossos dados ao avaliar a sensibilidade e especificidade de diferentes métodos de diagnóstico para a detecção do VFA. Os pesquisadores analisaram amostras de pacientes suspeitos de febre amarela, incluindo amostras de sangue, urina e saliva, e entre os métodos avaliados estava a RT-qPCR, amplamente reconhecida como uma técnica sensível e específica para a detecção do material genético viral.
Nossos resultados demonstraram um índice de eficiência e especificidade acima de 95% na detecção do VFA em amostras vacinais e selvagens, o que está em concordância com o estudo de Domingo et al.16, que também enfatizou a alta performance da RT-qPCR na detecção desse vírus. Essa concordância entre os dados obtidos em nosso estudo e os dados da literatura reforça a validade e confiabilidade de nossa abordagem.
É importante ressaltar que a detecção do VFA em amostras vacinais e selvagens desempenha um papel crucial no monitoramento da circulação viral e na identificação de surtos. Nossos resultados contribuem para o avanço do conhecimento nesta área, demonstrando a eficiência do método de RT-qPCR na detecção das diferentes cepas do VFA.
Nossos resultados revelaram uma eficiência e confiabilidade dos resultados em 90% para a detecção do VCHIK em amostras de parafina por meio da técnica de RT-qPCR. Esses dados são consistentes com estudos anteriores, como os mencionados por Silva et al.17 e Pialoux et al.18, que discutem a epidemiologia e os mecanismos de replicação do VCHIK.
Embora nossos resultados demonstrem uma eficiência ligeiramente menor em comparação com os estudos citados, é importante considerar que cada estudo possui suas particularidades, incluindo as características das amostras utilizadas, protocolos experimentais e condições específicas de detecção do vírus.
Apesar dessa diferença, é encorajador observar que nossos resultados estão alinhados com a literatura existente, que destaca a sensibilidade e especificidade da RT-qPCR na detecção do material genético do VCHIK em diferentes tipos de amostras. Essa concordância reforça a validade e a confiabilidade da nossa abordagem.
5 CONCLUSÕES
Em conclusão, o uso da técnica de RT-qPCR para a detecção de arbovírus em amostras de parafina apresenta benefícios significativos, permitindo análises retrospectivas e fornecendo informações valiosas para estudos epidemiológicos. Nossos resultados foram consistentes com estudos anteriores, demonstrando a eficiência e confiabilidade da RT-qPCR na detecção dos vírus da Dengue, Zika, Febre Amarela e Chikungunya em amostras de parafina. Nossa abordagem permitiu a identificação de sorotipos específicos, fornecendo insights importantes para compreender a dinâmica de transmissão, padrões sazonais e geográficos de infecção, e facilitando o desenvolvimento de estratégias de controle e prevenção mais eficazes. Esses resultados contribuem para o avanço do conhecimento sobre essas doenças e reforçam a validade e confiabilidade de nossa metodologia.
REFERÊNCIAS
- Guzman MG, Harris E. Dengue. Lancet. 2015 Jan 31;385(9966):453-65.
- Thammasonthijarern N, Puangmanee W, Sriburin P, Injampa S, Chatchen S, Phumirattanaprapin W, et al. Human Heavy Chain Antibody Genes Elicited in Thai Dengue Patients during DENV2 Secondary Infection. Jpn J Infect Dis. 2020 Mar 24;73(2):140-7.
- Vicenti I, Dragoni F, Giannini A, Giammarino F, Spinicci M, Saladini F, et al. Development of a Cell-Based Immunodetection Assay for Simultaneous Screening of Antiviral Compounds Inhibiting Zika and Dengue Virus Replication. SLAS Discov. 2020 Jun;25(5):506-14.
- Zhou Y, Mammen MP, Klungthong C, Chinnawirotpisan P, Vaughn DW, Nimmannitya S, et al. Comparative analysis reveals no consistent association between the secondary structure of the 3’-untranslated region of dengue viruses and disease syndrome. J Gen Virol. 2006 Sep;87(Pt 9):2595-603.
- Sam S-S, Omar SFS, Teoh B-T, Abd-Jamil J, AbuBakar S. Review of Dengue hemorrhagic fever fatal cases seen among adults: a retrospective study. PLoS Negl Trop Dis. 2013 May 2;7(5):e2194.
- Araújo PA, Freitas MO, Chiang JO, Silva FA, Chagas LL, Casseb SM, et al. Investigation about the Occurrence of Transmission Cycles of Arbovirus in the Tropical Forest, Amazon Region. Viruses. 2019 Aug 22;11(9).
- Lopez-Jimena B, Bakheit M, Bekaert M, Harold G, Frischmann S, Fall C, et al. Development and Validation of Real-Time RT-LAMP Assays for the Specific Detection of Zika Virus. Methods Mol Biol. 2020;2142:147-64.
- Thompson R, Martin Del Campo J, Constenla D. A review of the economic evidence of Aedes-borne arboviruses and Aedes-borne arboviral disease prevention and control strategies. Expert Rev Vaccines. 2020 Feb;19(2):143-62.
- Reed LJ, Muench H. A simple method of estimating fifty per cent endpoints. Am J Epidemiol. 1938 May;27(3):493-7.
- Johnson BW, Russell BJ, Lanciotti RS. Serotype-specific detection of dengue viruses in a fourplex real-time reverse transcriptase PCR assay. J Clin Microbiol. 2005 Oct;43(10):4977-83.
- Gustafsson RKL, Engdahl EE, Fogdell-Hahn A. Development and validation of a Q-PCR based TCID50 method for human herpesvirus 6. Virol J. 2012 Dec 18;9:311.
- Jonsson N, Gullberg M, Lindberg AM. Real-time polymerase chain reaction as a rapid and efficient alternative to estimation of picornavirus titers by tissue culture infectious dose 50% or plaque forming units. Microbiol Immunol. 2008 Dec 18;53(3):149-54.
- Guerra MJ, Ferreira CSS, Beraldo KRF, Kimura LM, Takahashi JPF, Salas-Gómez D, et al. One-step Multiplex Real-time RT-PCR for Molecular Detection and Typing of Dengue Virus Infection From Paraffin-embedded Tissues During the Brazilian 2019 Outbreak. Appl Immunohistochem Mol Morphol. 2021 Feb 1;29(2):158-62.
- Corchuelo S, Gómez CY, Rosales AA, Santamaria G, Rivera JA, Saad EP, et al. CISH and IHC for the Simultaneous Detection of ZIKV RNA and Antigens in Formalin-Fixed Paraffin-Embedded Cell Blocks and Tissues. Curr Protoc. 2021 Dec;1(12):e319.
- Reagan-Steiner S, Simeone R, Simon E, Bhatnagar J, Oduyebo T, Free R, et al. Evaluation of Placental and Fetal Tissue Specimens for Zika Virus Infection – 50 States and District of Columbia, January-December, 2016. MMWR Morb Mortal Wkly Rep. 2017 Jun 23;66(24):636-43.
- Domingo C, Patel P, Yillah J, Weidmann M, Méndez JA, Nakouné ER, et al. Advanced yellow fever virus genome detection in point-of-care facilities and reference laboratories. J Clin Microbiol. 2012 Dec;50(12):4054-60.
- Silva LA, Dermody TS. Chikungunya virus: epidemiology, replication, disease mechanisms, and prospective intervention strategies. J Clin Invest. 2017 Mar 1;127(3):737-49.
- Pialoux G, Gaüzère B-A, Jauréguiberry S, Strobel M. Chikungunya, an epidemic arbovirosis. Lancet Infect Dis. 2007 May;7(5):319-27.
* Graduanda em Biomedicina; Escola Superior da Amazônia, Belém, Pará, Brasil.
† Mestre em Virologia; Instituto Evandro Chagas, Ananindeua, Pará, Brasil
‡ Doutora em Neurociência, UFPA, Belém, Pará, Brasil.
§ Doutor em Virologia; Instituto Evandro Chagas, Ananindeua, Pará; Universidade Federal do Pará (UFPA), Belém, Pará, Brasil